이번 포스트에서는 Calcium Imaging과 관련된 segmention을 다룬 DISCo에 대해 공부한 것을 정리해보고자 한다.
Abstract:
칼슘 이미징은 신경생리학에서 중요한 도구 중 하나이다. 이를 통해 단일 세포 해상도에서 수백 개의 세포들의 신경적 활동을 병렬적으로 관찰할 수 있기 때문이다. 칼슘 이미징을 통해 얻은 데이터를 사용하기 위해 녹화된 영상으로부터 개별 세포들의 활동을 추출해야 한다. 이를 위해 이 논문에서는 DISCo를 제안한다.
DISCo는 픽셀 간의 상관관계를 계산하여 기록의 시간 정보를 계산적으로 효율적인 방식으로 사용하고 이를 형상 기반 정보와 결합하여 활성 및 비활성 셀을 식별한다. 처음에는 두 개의 픽셀이 하나의 세포에 속하는지 예측하는 것을 학습한다.
이를 통해 NP-hard correlation 군집 문제를 최근에 제안된 greedy algorithm을 통해 대락적으로 해결할 수 있다.
1. Introduction
칼슘 이미징은 단일 세포 해상도 수준에서 많은 신경 단위의 활동을 관찰할 수 있는 현미경검사 기술이다. 이 기술을 통해 뇌 안의 신경망의 생성과 상호작용에 대한 연구를 할 수 있다. 여기서 데이터는 연속적인 이미지로 다수의 세포들의 움직임에 따라 발광도가 달라지는 모습을 고정된 위치에서 관찰하여 얻는다. 칼슘 이미징 비디오에서 세포들의 위치를 추출하는 것은 핵심적인 부분이지만 아직 이 데이터에 대한 분석에서는 해결되지 않은 문제이다.
칼슘 이미징 비디오에서 활용할 segmentation 기법의 개발과 다를 접근법들과의 유의미한 비교를 가능하게 하기 위해 Neurofinder public benchmark가 추진되었다. Neurofinder challenge는 학습에 사용할 라벨이 있는 19개의 칼슘 이미징 비디오와 9개의 테스트 비디오로 구성되어있다. 이 둘의 데이터는 모든 5개의 데이터셋 시리즈(00,01,02,03,04)로 군집화 될 수 있다. 각각의 데이터셋은 다른 화경과 라벨링 기법의 차이, 그리고 ground truth가 active cell로 이루어져 있는지, non-active cell로 이루어져 있는지 아니면 이 둘이 함께 있는지와 같은 차이를 가지고 기록되었다.
이 논문에서는 DISCo를 제안하는데, 이는 딥러닝 모델의 장점과 SOTA한 instance segmentaion algorithm을 결합하여 cell instance를 직접적으로 추출할 수 있다고 한다. 추가적으로 픽셀 간의 segment-wise correlation을 계산함으로써 칼슘 이미지로부터 시간적 정보를 사용한다. 이런 시간적 정보는 shape-based 정보와 결합되는데 이는 하나의 정보만 사용하는 다른 방법들과 비교했을 때 큰 우위를 가지게 한다.
2. Related Work
칼슘이미징 데이터로부터 세포를 추출하기 위해서 현존하는 알고리즘들은 NMF(Non-negative matrix Factorization), clustering, dictionary learning, 그리고 딥러닝에 기반을 두고 있다.
UNet2DS와 Conv2D는 summary images고도 불리는 데이터를 입력으로 사용하는 하는 딥러닝 모델이다. 이러한 summary images들은 각각의 픽셀이 시간에 걸친 평균 사영을 담고 있다. 이는 모든 시간적 정보들이 소실되었다는 것을 의미한다. 그 결과, 이러한 접근은 많은 active cell을 담고 있는 데이터셋(01,02,04)에 대해서는 경쟁력이 없다.
반면에 HNCcorr는 이런 데이터셋에서 active cell 들을 잘 탐지한다. 왜냐하면 HNCcorr는 correlation space에서 픽셀 간의 거리에 기반한 clustering을 사용하기 때문이다. 이 correlation space 변화하는 신호를 가진 픽셀들은 배경 픽셀들과 분리가 잘된다. 하지만 약하거나 지속적인 활동을 보이는 셀들의 픽셀들은 잘 구분하지 못한다.
딥러닝 모델 중 하나인 STNeuroNet 과 이 모델의 개발 단계인 3 dCNN은 모든 테스트 셋에서 3dCNN을 사용하여 좋은 F1-score를 얻었었다. 이러한 모델의 연산 비용이 크기 때문에 짧은 시간적 배치로 모델을 사용하여 전체 비디오에서 세포의 위치를 얻었고, 다른 배치에서의 출력은 후처리를 통해 병합했다. 다른 모든 모델처럼 STNeuroNet과 3dCNN은 전경과 배경 예측만을 제공하고 개별의 cell instance를 추출하기 위해 후처리가 필요하다. STNeuroNet은 추가로 ABO 데이터셋과 수작업으로 정제한 ground truth를 학습했다. 반면에 3 dCNN Neurofinder challenge에 사용한 데이터셋과 ground truth만 사용했다.
3. Method
DISCo는 앞서 언급한 대로 픽셀 간의 segment-wise correlation을 사용하여 시간적 정보를 추출한다. 이러한 시간적 정보는 summary image로부터 shape-based 정보와 결합하여 affinities로 변환된다. 그리고 이 affinities는 개별 세포들을 추출하고 분리하는 sota 한 segmentation algorithm에 사용된다.
DISCo는 시간적으로 영상을 픽셀 간의 correlation을 계산할 segment로 분리한다. correlation coefficient의 segment-wise 연산은 신경적 활동 데이터 분석에 성공적으로 쓰여왔다. 이 방법의 장점은 SNR과, 픽셀의 시간적 변화에 대한 fine-grained 정보의 향상이다. 다수의 segment에 대한 correlation을 계산하는 것은 영상 텐서의 시간적 차원을 감소시키는 것을 말한다. 그러므로 저자들은 작은 3dCNN을 이용하여 다른 segment로부터 정보를 합친다. 이와 더불어 summary image는 전체 영상 시간에 걸친 각각의 픽셀의 평균 사영을 이용하여 계산된다. Summary image는 통합된 정보와 결합되어 2번째 네트워크에 시간적, shape-based 정보를 제공한다. 두 번째 네트워크는 이 입력값을 affinities에 매핑한다. 마지막 단계에서는 예측된 afiinities로부터 undirected, edge-weighted graph를 구성하고, 개별 세포들은 이 그래프들은 나누면서 추출되고 분리된다. Pixel-wise affinities와 더불어 신경망은 clustering을 하기전에 직접적으로 그래프에서 배경 픽셀을 제거하는 데 사용하는 전경-배경 예측을 제공한다.
3.1 Temoporal Information from correlations
세포들의 발광 변화와 배경 픽셀의 발광 변화가 큰 차이를 보이므로, 칼슘 이미지의 시간적 정보를 사용하는 것은 세포를 탐지하는데 큰 도움이 된다. 게다가 시간적 정보 없이는 인접하거나 겹친 세포들을 올바르게 분리하는 것이 어렵다.
이런 이유 때문에 저자들은 칼슘 이미지 영상 내 시간적 정보를 correlation의 형태로 사용한다. 이 연구에선 Pearson correlation을 사용한다. 왜냐하면 이미지와 긴 시계열 데이터에 대해 연산이 빠르고 효율적이기 때문이다. 덕분에 신경망을 학습하는 동안 correlation을 온라인(실시간)으로 계산할 수 있고 이는 더 넓은 범위의 데이터 증강 단계를 가능하게 한다.
저자들은 전체 영상에 걸쳐 두 픽셀 간의 correlation을 계산하지 않고 먼저 영상을 10개의 segment로 나누고 이 나눈 segment별로 correlation을 계산했다. 저자들은 segment-wise correlation만 사용한 것이 아니라 다른 segment의 정보를 통합하기 위해 CNN을 함께 사용했다. segment-wise correlation는 전체 영상에 걸친 correlation 보다 더 정교한 시간적 정보를 제공한다. 더 나아가 단순히 인접한 픽셀끼리 correlation을 구한 것이 아니 주변에 있는 최대 15개의 픽셀 간의 correlation을 계산하여 사용했다.
3.2 Deep Neural Network
Networks
사용된 신경망은 두 개의 파트로 나뉜다. 첫 번째 파트는 segment-wise correlation의 정보를 통합하는 3D convolution이 있는 CNN이다. 이 신경망의 출력은 summary images와 2DUNet 구조의 두 번째 신경망으로 전달된다. 이 신경망의 출력은 픽셀 간 예측된 affinities와 전경-배경 예측이다. 이 두 신경망은 두 번째 신경망의 출력에 Sørensen Dice loss를 적용하여 함께 학습되었다.
Training
correlations과 summary image는 channel-wise 하게 평균이 0이고 단위 분산을 가지게끔 정규화하였다. 학습을 위해서 ground truth를 픽셀과 전경-배경 라벨 값 간의 affinities로 변환했다. 그리고 모든 출력에 channel-wise Sørensen Dice loss를 적용했다. 왜냐하면 해당 loss가 affinities를 학습하는데 성공적이었고 전경-배경간에 존재하는 큰 class imbalance를 잘 처리하기 때문이다. 적합한 하이퍼 파라미터를 찾기 위해, 모든 영상을 각각 75%는 학습용, 25%는 검증용으로 분할하여 파라미터들을 테스트하고 validation loss를 기반으로 best setting 값을 정의했다. 최종으로 사용한 하이퍼 파라미터는 다음과 같다.
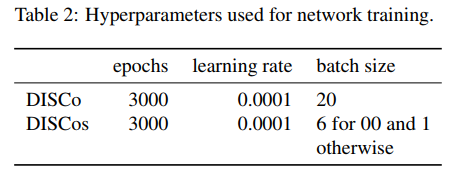
Data Augmentation
Neurofinder 학습 데이터가 19개의 비디오만 제공되므로, 성공적인 학습을 위해선 data augmentation이 필수다. Data Augmentation은 시간적, 공가적 차원 모두에 사용했다. Correlation을 계산하기 전 신호들의 노이즈를 줄이기 위해 영상의 시간 변화에 따라 max-pooling을 수행했다. 학습을 위해선 3개와 9개의 프레임 간의 max-pooling 커널의 시간적 길이에 변화를 줬다. 추정을 위해선 max-pooling 커널의 길이를 5개로 고정했다. 학습 동안 correlation을 계산하기 전 영상을 임의로 10개의 부분으로 잘라 shuffle 하였다. 공간적 차원을 위해서는 random flips와 rotation을 이용했다. 추가로 사이즈가 128 x128인 이미지의 random crop 하여 학습했다. 그리고 각각의 crop은 적어도 한 개의 세포를 포함하도록 했다.
3.3 Instance segmentation
실제 세포 객체를 추출하기 위한 최종 단계로 GASP(Generalized Algorithm for Signed graph Partitioning)을 사용한다. HNCcorr에 사용되는 HNC(Hochbaum's normalized cut)과 Conv2D와 STNeuroNet의 후처리를 위한 watershed 알고리즘과 반대로 GASP는 partitioning을 정지하기 위한 threshhold나 seed나, 사전에 정의된 군집의 개수가 필요하지 않다.
GASP는 signed graph $G = (V, E, W )$를 분할하기 위해 고안되었다. $V$ 는 노드를, $E$ 는 에지를, 그리고 $W$는 가중치를 의미한다. 이 연구에서 $V$는 칼슘 이미징 영상의 pixel에 해당하고 엣지들의 구조는 아래의 그림처럼 사전에 정의되었다.

칼슘 이미징에서의 세포들은 비교적 작으므로, 작고 인접한 세포들의 분리를 가능하게 하기 위해 5픽셀 거리까지만 픽셀들을 연결했다. 높은 가중치를 가지는 엣지는 노드들이 동일한 군집에 속할 경향성을 의미한다. 반대로 높은 음수의 가중치는 노드들이 다른 군집에 속할 가능성이 높은 것을 의미한다. GASP알고리즘은 한 노드가 자신만의 군집에서 시작하여 반복적으로 인접한 군집들을 병합한다. 여기서 인접한 군집의 의미는 적어도 하나의 연결하는 엣지가 존재한다는 것을 뜻한다. 그리고 두 군집 간의 상호작용이 positive 한 것을 의미한다. 세포와 배경간 잘못된 병합과 모든 배경 객체를 제거하기 위해 분할 전에 그래프에서 배경과 연결되는 모든 엣지들을 제외한다. 하나의 픽셀이 배경과 연결되었는지의 여부는 전경-배경 예측에 기반한다. 배경이라고 예측된 모든 픽셀들은 그래프에서 제외한다. 이 과정은 결과를 조금 향상시키고 instance segmentation 과정의 속도를 한층 더 빠르게 했다. 마지막 단계로 아주 작은 배경 segment를 제거하기 위해 최종 결과에서 25픽셀 이하의 모든 객체들을 제외하는 간단한 threshhold를 사용했다.
4. Experiments and Results
연구진은 DISCo를 앞서 언급한 하이퍼 파라미터를 적용하여 Neurofinder 학습 셋으로 학습했다. 그리고 9개의 테스트 비디오에 대한 평균 F1 스코어로 결과를 평가했다. DISCo는 summary image기반의 UNet2DS와 Conv2D의 성능을 뛰어넘는 결과를 보였다. 더 나아가 HNCcorr과 같이 correlation에 기반한 모델의 결과보다 훨씬 더 좋은 결과를 보였다.

Lesion Study
correlation과 summary image를 둘 다 사용하는 것의 이점을 강조하기 위해 DISCo를 아래와 같이 input을 다르게 하여 실험했다.
1) summary images + segment-wise correlation
2) 영상 전체에 걸친 summary images + segment-wise correlation
3) summary images only
4) correlation only
그리고 아래와 같은 결과와 같이 1번 (summary images + segment-wise correlation)의 결과가 가장 좋았다.

Training with only one Video
실제로 segmentation을 하고자 하는 칼슘 이미징 데이터는 Neurofinder challenge에서 제공하는 데이터와 다를 수 있다. 이 문제에 접근할 수 있는 한 가지 방법은 모델이 일반화를 잘하기를 바라면서 최대한 많은 데이터셋으로 학습을 하는 것이다. 하지만, 굉장히 다양한 칼슘 인디케이터들과, 녹화 환경, 관찰하고자 하는 뇌의 부위 등 다양한 조건들을 단일 모델로 처리할 수 있을지가 의문이다. 더 나아가서 연구 주제에 따라서 모델이 active cell 만 탐지하길 바랄 수도 있고 active cell과 inactive cell을 동시에 탐지하기를 원할수 있다.
이런 이유로 연구진은 하나의 영상으로 학습한 모델을 가지고 테스트를 진행하기로 했다. 새로운 환경에서 촬영한 영상을 뇌공 학자가 직접 라벨링 한다면, 이 영상은 똑같은 환경에서 segmentation을 필요로 하는 데이터를 위해 모델을 학습하는 데 사용할 수 있다고 한다. 그리고 그 결과는 다음과 같다.

Precision and Recall
F1스코어 관점에서 DISCo는 좋은 성능을 보인다. 하지만, precision과 recall 관점에서 몇몇의 데이터 셋에 대해서 DISCo는 precision에 비해 비교적으로 낮은 recall 값을 보이고 다른 데이터 셋에서는 반대의 경향을 보이기도 한다. 이 현상에 대한 원인은 아직 밝히지 못했지만, 추후에 다른 데이터셋의 결과와 특성을 분석하여 모델의 이런 현상의 원인을 찾아야 한다고 한다.
5. Summary and Conclusion
이 논문의 저자들은 칼슘 이미징 영상 segmentation의 새로운 방법을 제시한다. 이들은 딥러닝 모델을 사용하지만, 이전의 연구와 다르게 단순히 입력으로 shape-based summary image에만 의존하지도 않고 연산적으로 비용이 큰 3dCNN 또한 사용하지 않는다. 그 대신 더 빠르고 효율적인 framework를 제안한다. 입력으로는 pixel과 summary image를 함께 사용한다. 이 방식을 통해 active cell뿐만 아니라 약하거나 거의 동일한 신호를 보이는 세포들을 탐지할 수 있다. 그리고 segment-wise 하게 correlation을 구하는 방법으로 더 정교한 시간적 정보를 얻을 수 있다고 한다. 또 다른 novelty는 연구에 사용된 딥러닝 모델이 전경과 배경을 예측하는 것뿐만 아니라 pixel 간의 affinities를 예측할 수 있다는 것이다. 이는 직접적으로 신경망의 결괏값에 instance segmentaion을 적용할 수 있게 되어 개별 세포들을 추출할 수 있게 한다.
Future work로는 왜 몇몇의 데이터에서는 precision 보다 recall이 낮고 다른 데이터에선 반대의 현상이 나타나는지 분석을 해보는 것도 흥미로운 주제라고 한다.
'AI' 카테고리의 다른 글
| [ERROR] CUDA error: CUBLAS_STATUS_INVALID_VALUE 해결법 (0) | 2023.04.26 |
|---|---|
| [파이토치] 모델 저장 및 불러오기 (1) | 2022.10.07 |
| [파이토치]최적화 (1) | 2022.10.06 |
| [파이토치]자동미분_AUTOGRAD (0) | 2022.09.30 |
| [파이토치]신경망 구성 (0) | 2022.09.29 |




댓글